抗生素,抗生素耐药性与环境
微生物自身会产生抗生素,抑制其他竞争性微生物的生长。为了生存,微生物通过遗传和生化机制进化出了抗生素抗性, 并通过频繁的遗传物质互换在种群间广泛传播。然而,自1940年代以来,人类开发出了新型抗生素,使用量逐年增大 。在15年间,全球抗生素消费量增长了65%,主要集中于中低收入国家。目前,全球性的抗生素大规模使用已成为一个主要的公众健康隐患。 某些对人和动物具有致病性的细菌已经对制药工业开发的大多数抗生素分子产生了耐药性。除此之外,施用于人和动物的抗生素,以及来源于宿主的耐药菌已在环境中广泛传播。 细菌正向抗生素耐药性能力增加的方向进化。 环境、动物或人类宿主中的菌群之间的基因交换永远存在。 因此,对抗生素耐药性的斗争必需全面考虑。抗生素对不同宿主的影响难道不应该被考虑在内吗?
1. 细菌耐药性:一个全球性的健康威胁
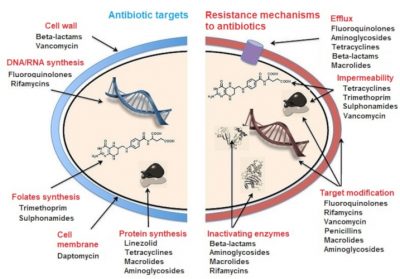
[来源: Creative Commons Attribution 2.0 Generic License.]
抗生素靶点 抗生素耐药性机制
细胞壁:β-内酰胺类 万古霉素类 流出:氟喹诺酮类 氨基糖甙类 四环素类
β-内酰胺类 大环内酯类
DNA/RNA合成:氟喹诺酮类 利福霉素类
叶酸合成:甲氧苄氨嘧啶类 磺胺类 不渗透性:四环素类 甲氧苄氨嘧啶类 磺胺类万古霉素类
细胞膜:达托霉素类 靶点修饰:氟喹诺酮类 利福霉素类 万古霉素类 青霉素类 大环内酯类 氨基糖甙类
蛋白质合成:利奈唑胺类 四环素类 大环内酯类 氨基糖甙类 灭活酶:β-内酰胺类 氨基糖甙类 大环内酯类 利福霉素类
一组抗生素耐药性对公众健康风险影响的数据值得研究人员警惕:2015年,全球每天消耗423亿剂的抗生素 。耐药性指某些细菌耐受一种或多种抗生素作用的能力,由细菌基因组中的某些基因编码。 对于特定抗生素,细菌的耐受性主要包括四种生物化学机制:
- 细菌膜对该抗生素的抗渗性;
- 细菌对渗入抗生素的外排;
- 通过对作用靶点的定量或定性修饰,降低抗生素与细菌靶点的亲和力;
- 利用细菌酶使抗生素失活。
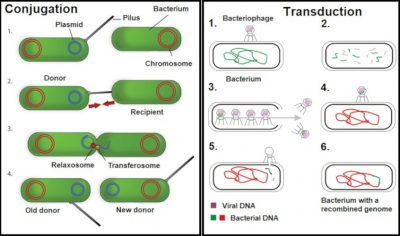
[来源: 左侧翻译自By derivative work: Franciscosp2 (talk) Bacterial_Conjugation_en.png: Mike Jones (Bacterial_Conjugation_en.png) [CC BY-SA 2.5], via Wikimedia Commons ; 右侧翻译自By Reytan with modifications by Geni & Toony (common Image: Transduction (genetics)en.svg) [Public domain], via Wikimedia Commons.]
接合:1.质粒 菌毛 细菌 染色体2.供体细菌 受体细菌3.松弛体 转染体4.新的供体 旧的供体 转导:1噬菌体 细菌5.病毒DNA 细菌DNA 6.重组基因组的细菌
图1概述了抗生素的作用靶点和细菌对这些分子的耐药性机制。
某些种类细菌的耐药性机制稳定,并影响大多数菌株:我们称之为天然耐药性。相反,当某类对抗生素敏感细菌的特定菌株通过遗传机制获得抗性时,即会产生获得性耐药性 。获得新的抗生素耐药性可能与突变相关,并受抗性基因的特性和表达水平影响(参见Genetic Polymorphism and Variation)。也可能是获得新的耐药基因的结果。事实上,移动基因元件[1](质粒、转座子、整合子等)可以在相同或不同种类的细菌之间交换。这些交换通过水平基因转移机制[2](接合、转化、转导、转座)进行(图2和3)。所有存在于致病性或非致病性微生物中的抗生素耐药性基因构成了抗性组。
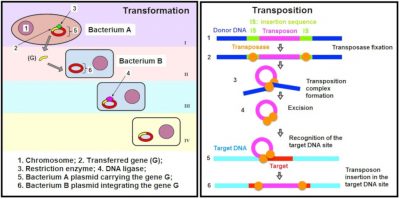
[来源:左边改编自By Sprovenzano15 [CC BY-SA 3.0], from Wikimedia Commons; 右边改编自 Alana Gyemi [CC BY-SA 4.0], from Wikimedia Commons.]
转化:细菌A,细菌B 1.chromosome染色体;2.transferred gene(G)转移的基因(G);3.restriction enzyme限制性内切酶;4.DNA ligase DNA连接酶;5.bacteriumA plasmid carrying the gene G 携带基因G的细菌A质粒6.bacteriumB plasmid integrating the gene G整合基因G的细菌B质粒
转座:insertion sequence插入序列; donor DNA供体DNA;transposon转座子; transposase转座酶; transposase fixation转座酶固定; transposition complex formation转座复合物的形成; excision切除; recognition of the target DNA site识别靶DNA位点; target DNA靶DNA; transposon insertion in the target DNA site转座子插入靶DNA位点.
在过去的80年里,抗生素的大量使用导致了新的抗生素耐药性机制的选择和种间转移。在同一种细菌中的耐药性的累积导致多耐药性(对多个抗生素家族产生耐药性,MDR),甚至是完全耐药性(对所有可用抗生素产生耐药性)。这两种情况会造成治疗僵局。某些经常涉及人体病理,且对多种抗生素具有耐药性的细菌种类被统称为超级耐药菌(ESKAPE)[3],包含屎肠球菌、金黄色葡萄球菌、肺炎克雷伯菌、鲍曼不动杆菌、铜绿假单胞菌、肠杆菌及最近发现的其他肠杆菌科细菌(非大肠杆菌)(表1)。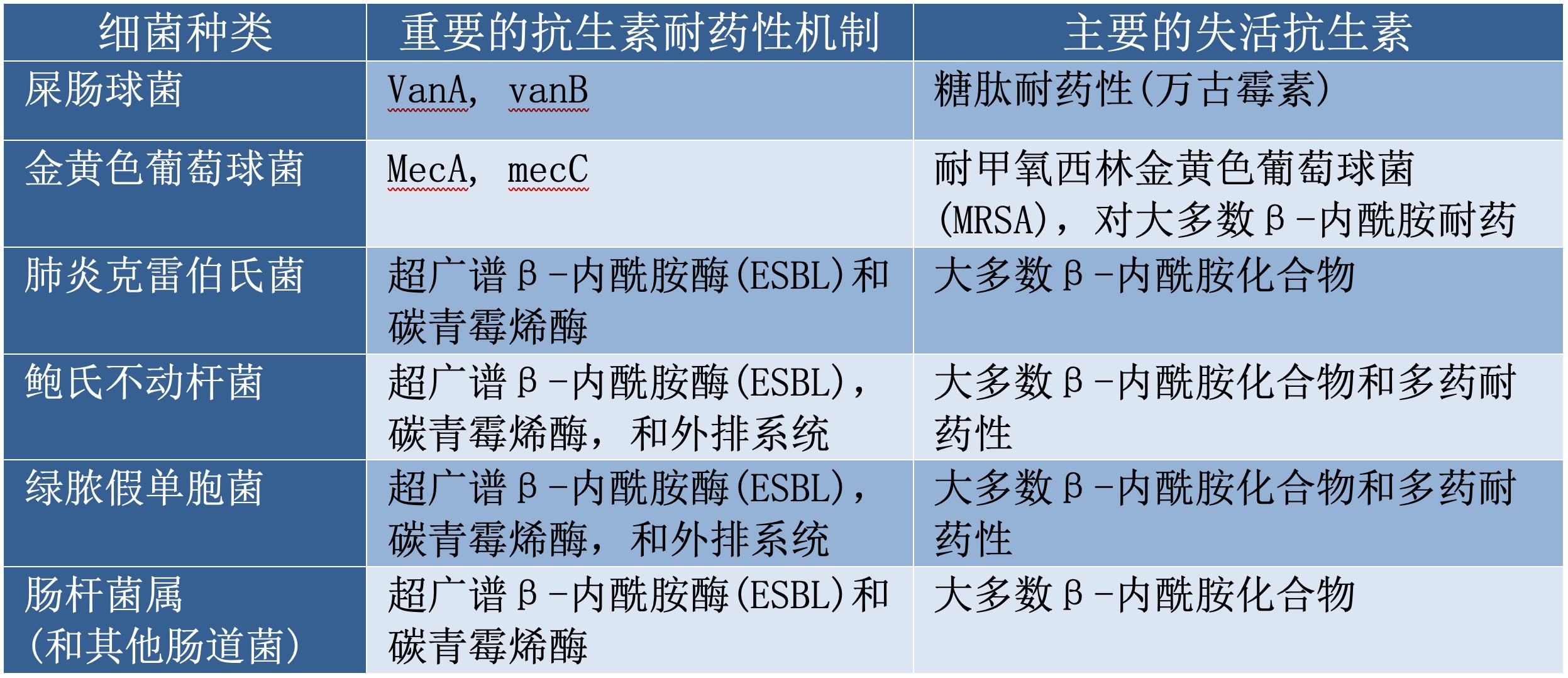
当前,细菌耐药性是一个重大的全球公共卫生问题, 需要依靠国际(包括世卫组织)和国家控制计划的进程。尽管已在人和动物宿主中鉴定了抗生素耐药性,但直到最近才认识到环境在耐药性的出现和传播中起到的作用。
2. 环境:古老的抗生素耐药性宿主
许多环境微生物 (尤其是细菌和真菌)可以自发地产生抗生素,因此赋予它们较其他环境物种具有选择性生长的优势。这些微生物具有抵抗自产抗生素的耐药性机制的基因编码。它们可以把这些耐药性基因传递给后代(垂直传递)[4]。不产生抗生素的微生物也可能携带抗生素抗性基因或从头获得它们。在细菌中,相同或不同物种的个体之间存在许多遗传交换(水平传递)。 生活在环境中细菌之间的耐药性传播可能从很久以前(几十亿年)就开始了,并持续至今[5]这些耐药性可以从环境细菌传播到人类和动物身上的细菌。例如,最近出现的CTX-M型广谱b-内酰胺酶(ESBLs)或对氟喹诺酮类药物(qnr基因)产生耐药性的新机制[6]。
3. 人造抗生素的环境释放
20世纪初的重大标志是发现并大量生产天然抗生素(从微生物中提取)或通过化学合成获得。天然抗生素包括b-内酰胺类抗生素(包括青霉素)、氨基糖苷类、四环素类和大环内酯类药物。其中大部分天然分子经过化学修饰,成为半合成抗生素或最近的合成抗生素。磺胺类和喹诺酮类药物是人类直接合成的分子。从20世纪40年代到现在,人和动物使用的抗生素呈指数式增长,相当于向环境释放了数百万吨的抗生素。抗生素对环境的污染主要有三种来源:
- 抗生素工业生产厂向水环境的排放;
- 饲养家畜所使用的抗生素;
- 用过抗生素的人。
3.1. 抗生素生产工厂
抗生素生产工厂通过废水排放了大量的抗生素。 在大量生产抗生素的国家,特别是在欧洲、美国、中国和印度,目前在环境污染方面的监管限制是不够的。某些工厂的废水中可检测到的抗生素浓度超过1毫克/升[7]。这种浓度对环境微生物菌群有显著影响。此外,环境中抗生素的广泛传播会污染地下水。
3.2. 兽医使用的抗生素
家畜的抗生素施用(很少包含野生动物)约占抗生素总产量的约60% ,并且还在持续增加[8]。全世界每年施用于家畜、低地动物和水产养殖的抗生素超过10万吨[9]。最常用的是四环素,其次是青霉素类和磺胺类药物。然而,b-内酰胺类抗生素、氨基糖苷类、氯霉素类、大环内酯类和糖肽类药物也同样受到关注 。人和动物所使用的抗生素属于相同的家族,因此也会由于相同的 耐药性机制而失活。抗生素用于治疗和预防家畜的传染病(抗生素预防),或者作为食物补充剂。为了治疗和预防细菌感染,即使只有少量家禽患病,抗生素常被添加到饮用水或整个农场的饲料中 。系统地使用抗生素作为饲料补充剂 (尤其是家畜、家禽、鱼等)是为了提高牲畜的产量 (例如,就生产肉的量而言 )。
数十年来,这种以盈利为目的使用抗生素的做法一直占绝大多数 。目前这种做法在欧洲已被禁止, 但在许多国家仍然存在。这被认为是动物消化道内定植细菌(如大肠杆菌)产生耐药性的主要原因。大多数从这些动物中筛选出来的耐药菌可以通过接触或被污染的食品传播给人类。 此外,动物服用的抗生素也在其粪便中保有活性。这些农场的废水 通常直接排放到水环境中,或未经事先处理就用于作物灌溉。
3.3. 人类对抗生素的使用
人类使用抗生素治疗或预防传染病(抗生素预防)。全球人口每年消耗数百亿单位剂量的抗生素。b-内酰胺类抗生素(青霉素类、头孢菌素类、碳青霉烯类)是目前使用最广泛的抗生素(约占人类消耗量的60%)。四环素类、大环内酯类和氟喹诺酮类药物的用量也很大。大约80%的抗生素在社区中使用,20%在医院使用。然而,由于广谱抗生素的使用和选择性耐药菌人传人的高风险 [10],因此医疗保健机构在细菌耐药性传播方面的角色很重要。一些开给患者的抗生素处方药没有被使用,因此和我们的日常废物一起释放到环境中。
更为重要的是,人体吸收的大部分抗生素通过尿液和粪便排出时仍具有活性。在发展中国家,这些抗生素被直接分散在大地-水环境中 。在拥有废水处理基础设施的国家,大部分的抗生素会被废水处理厂截留[11]。在一些处理厂的废水中可检测到的抗生素(b-内酰胺类、大环内酯类、四环素类、氟喹诺酮类等)浓度大约为mg/L级。这些抗生素被生物降解,被污水污泥吸附,或通过工厂废液排出。污泥可用作农田肥料,而污水被排入水环境(河流)。这两种情况下,活性抗生素将被释放到水陆环境中
- 环境中的抗生素残留浓度
在一些水和陆地环境中,可检测到的抗生素浓度介于纳克至微克每升水或每克土壤。一些地下水中也可测到类似的浓度。在污染后,这些抗生素会在环境中持续存在一段时间,从几天(如b-内酰胺抗生素)至几个月(如氟喹诺酮类和四环素类药物)。对于后者,如果环境污染频繁或持久,就会出现抗生素的累积现象。海洋环境中也可以检测到抗生素的残留。最终,有时在饮用水中可以检测到浓度在纳克每升水平的抗生素残留。
4. 人源和动物源的新型抗生素抗性基因的环境释放
用于人和动物的抗生素是筛选其共生菌群(其微生物菌群)中抗生素抗性最强的细菌种类和菌株。(参见Human microbiots: Allies for our health)。
通过促进基因突变和抗生素抗性的水平基因转移,抗生素还会在细菌SOS系统中诱导耐药性。这种现象在菌群丰富、细菌种类多达几千种的肠道菌群中尤为重要(图4)。
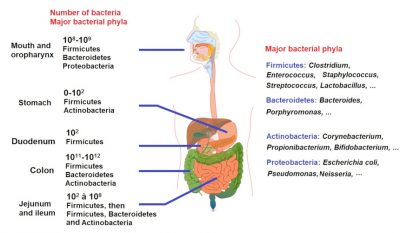
[来源: Adapted from By Mariana Ruiz, Jmarchn, Translated in French by Moez [Publicdomain], from Wikimedia Commons.]
Number of bacteria细菌数量; major bacterial phyla主要细菌门; mouth and oropharynx口腔和口咽; stomach胃; duodenum十二指肠; colon结肠; jejunum and ileum空肠和回肠;
Firmicutes厚壁菌门:梭菌,肠球菌,葡萄球菌群,链球菌,乳杆菌…
Bacteroidetes拟杆菌门:细菌,卟啉单胞菌…
Actinobacteria放线菌门:棒状杆菌,丙酸杆菌,双歧杆菌…
Proteobacteria变形细菌门:大肠埃希氏菌,假单胞菌,奈瑟菌…
人和动物向环境中传播构成其共生菌群的细菌,特别是携带抗生素抗性基因的细菌。有些种类的细菌可以在环境(水、土壤、被污染的物品等)中存活很长时间。由于人类活动,废水处理厂汇聚了大量废水 ,聚集了大量抗生素耐药菌及其抗性基因。严重污染的污泥常被用作农地化肥。净化后仍不达标的水被释放到环境中, 然而这些水仍然含有细菌和抗生素抗性基因。因此,污水处理厂是新的抗生素抗性基因造成环境污染的主要源头。
5. 环境中出现的新的抗生素抗性产物
水陆环境介质富含微生物,其多样性仅得到部分表征。近期宏基因组学研究导致许多新菌种的发现 。研究表明,超过99%的环境细菌物种不能用目前的方法培养[12]。
环境持续被人源或动物源抗生素抗性细菌、抗性基因编码以及人类制造的抗生素污染。 植物会吸收部分抗生素,尤其是被来自于废水处理厂含抗生素的污水或污泥灌溉的农作物。虽然环境中残留的抗生素浓度通常很低,但它们对天然微生物菌群的筛选压力日益增加。这种筛选压力促进了天然物种和污染物种之间抗生素抗性基因的水平交换,特别是对于在生物膜中丰富多样的细菌种群。这些微生物的天敌,即自由生存的原生动物(特别是变形虫),也促进了这些基因的交换。
总而言之,无论是在自然环境中的细菌菌群中,还是能够在这种环境中生存的人类和动物菌群中。 环境中已经具备新的抗生素抗性出现的条件。
6. 环境是人类和动物中新抗生素抗性的来源
来自水陆环境的微生物经常定植在人类和动物身上。这种定植可直接通过日常活动与环境接触而发生,或间接通过饮食、饮水,亦或被污染的物品而发生[13]。与人和动物共生的微生物菌群作为微生物屏障,可防御被一些机体缺乏适应性的外来物种感染。尽管如此,许多源自人类和动物的细菌物种仍可在环境中生存 ,并且能再次感染宿主。另一方面,环境微生物可以在人和动物的皮肤和粘膜中定植。这种定植可能是瞬时的、短期的(几小时或几天),或长期的(几周、几个月甚至几年)。在定殖期间,外源菌群和内源共生菌群之间的遗传交换会不断发生。由于消化道菌群的丰富性和多样性,并且许多环境微生物被摄入后在肠道内长时间停留,因此这些消化菌群的遗传交换尤为重要。
需要注意的是,耐药菌对抗菌剂同样会产生抗性,包括重金属(银、铜、汞等)和生物杀菌剂(乙醇、甲醛、洗必泰、三氯生、季铵盐 等。抗菌剂的环境污染会促使耐药菌的形成。两种现象可能有助于理解抗菌剂对耐药性菌株的筛选(或为抗菌剂抗性的抗生素选择):
- 抗生素和抗菌剂的抗性遗传机制由相同的基因载体(特别是质粒)携带;细菌暴露于抗生素或抗菌剂会筛选对这两类化合物都具有抗性的菌株,即为复合抗性;
- 相同的遗传机制(例如外排泵的基因编码)导致细菌对抗生素和抗菌剂同时产生抗性; 当这些分子中的一种或另一种存在时,会引起两类抗性的筛选,即为交叉抗性。
7. 综合展望:更好地理解与环境和集约化畜牧业相关的风险
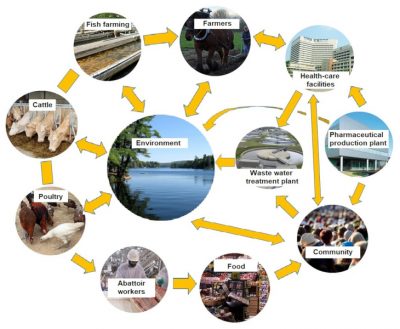
Cattle, fish farming, farmers, health-care facilities, pharmaceutical production plant, community, waste water treatment plant, environment, food, abattoir workers, poultry
牛,养鱼,农民,卫生保健机构,制药厂,社区,废水处理厂,环境,食品,屠宰场工人,家禽
抗菌药物耐药性是一个复杂且长期存在的现象,已成为人类和动物健康的一个主要问题,由三个因素共同作用 :1/几十年间抗生素的过度使用;2/基因组的高可塑性,并因此使得细菌能够适应选择压力; 3/环境、动物和人类:三个主要抗性基因库之间永远存在交换(图5) 。
大量而迅速的国际贸易 (人类、动物、食品)使这种情况恶化。环境受到来自于人源和动物源的耐药微生物以及残留抗生素的污染。这两类污染促进了新的抗生素抗性的出现以及细菌物种之间的耐药性转移。
由于人、动物和环境三者之间存在很强的相互作用,因而抗菌药物耐药性遗传机制的扩散广泛迅猛。与其他环境污染物(其他药物、化学品、重金属等)相比,这种污染具有能在人类和动物群体中潜在传播的特征。事实上,耐抗生素细菌在人与人、动物与动物和人与动物之间的传播不仅解释了这种污染的个体效应,也解释了其群体效应。在防治抗菌药物耐药性出现和传播的方法中,以下几点至关重要:
- 减少和优化抗生素的使用量;
- 避免或至少限制抗生素和携带抗性基因的细菌在环境中的传播,特别是重要的源头(医院、抗生素制造厂、集约型畜牧场、污水处理厂等);
- 提高发展中国家的卫生水平并开发水净化系统;
- 更好地监测抗生素及其抗生素抗性基因的环境污染[14]、[15]。
8. 要点
- 全球抗生素的消耗量是每天数十亿剂量,而且还在增加。
- 人类和动物使用的抗生素极大地改变了它们的共生微生物群落,并筛选出了对这些分子抗性越来越强的细菌菌株。
- 人类和动物致病菌的适应能力与其基因组可塑性有关,使得它们不断发展和交换新的抗菌药物耐药性机制。
- 人类和动物消耗的抗生素以及具有抗菌药物耐药性机制的细菌在环境中以活性形式分布。
- 环境是众多细菌种类和抗生素抗性基因的天然来源库,并且人源和动物源的污染使其不断丰富。
- 在人类、动物和环境之间不断的交换,会使抗生素抗性的出现和传播长久地持续下去。
- 有必要开发新的抗生素和新的治疗策略来抵御抗生素抗性,并减少和优化目前人类和动物的抗生素使用量。
参考资料及说明
封面图片:在测试抗生素敏感性之前,在选择性培养基上分离细菌菌落。[来源:Getty Images, royalty-free.]
[1] 一个可移动的遗传元件是基因组中位置不固定的部分。这些元素是由1983年获得诺贝尔生理学或医学奖的美国细胞遗传学家芭芭拉·麦克林托克发现的。它们种类繁多,包括质粒、转座子、整合子。
[2] 一个生物体整合来自另一个生物体的遗传物质而不是其后代的过程(也称为横向基因转移)。 基因工程的很大一部分由基因的人工水平转移组成。
[3] Pendleton JN, Gorman SP, Gilmore BF. (2013). Clinical relevance of the ESKAPE pathogens. Expert Rev Anti Infect Ther. 11(3):297‑308.
[4] Finley RL, Collignon P, Larsson DGJ, McEwen SA, Li X-Z, Gaze WH, et al (2013). The scourge of antibiotic resistance: the important role of the environment. Clin Infect Dis, 57(5):704-10.
[5] D’Costa VM, King CE, Kalan L, Morar M, Sung WWL, Schwarz C, et al (2011). Antibiotic resistance is ancient. Nature. 477(7365):457‑61.
[6] Cantón R. (2009). Antibiotic resistance genes from the environment: a perspective through newly identified antibiotic resistance mechanisms in the clinical setting. Clin Microbiol Infect. 15 Suppl 1:20-5.
[7] Larsson DGJ. (2014). Antibiotics in the environment. Ups J Med Sci. 119(2):108-12
[8] Singer AC, Shaw H, Rhodes V, Hart A. (2016). Review of Antimicrobial Resistance in the Environment and Its Relevance to Environmental Regulators. Microbiol Front. 7:1728.
[9] Lekshmi M, Ammini P, Kumar S, Varela MF. (2017). The Food Production Environment and the Development of Antimicrobial Resistance in Human Pathogens of Animal Origin. Microorganisms. 5(1).
[10] Hosein IK, Hill DW, Jenkins LE, Magee JT. (2002). Clinical significance of the emergence of bacterial resistance in the hospital environment. J Microbiol application, 92 Suppl:90S-7S.
[11] Le-Minh N, Khan SJ, Drewes JE, Stuetz RM. (2010). Fate of antibiotics during municipal water recycling treatment processes. Water Res. 44(15):4295-323.
[12] Nesme J, Cécillon S, Delmont TO, Monier J-M, Vogel TM, Simonet P. (2014). Large-scale metagenomic-based study of antibiotic resistance in the environment. Curr Biol, 24(10):1096-100.
[13] Zurek L, Ghosh A. (2014). Insects represent a link between food animal farms and the urban environment for antibiotic resistance traits. Appl About Microbiol. 80(12):3562-7.
[14] Pruden A, Larsson DGJ, Amézquita A, Collignon P, Brandt KK, Graham DW, et al (2013). Management options for reducing the release of antibiotics and antibiotic resistance genes to the environment. About Health Perspect. 121(8):878‑85.
[15] Laxminarayan R, Duse A, Wattal C, Zaidi AKM, Wertheim HFL, Sumpradit N, et al (2013). Antibiotic resistance-the need for global solutions. Lancet Infect Dis. 13(12):1057-98.
环境百科全书由环境和能源百科全书协会出版 (www.a3e.fr),该协会与格勒诺布尔阿尔卑斯大学和格勒诺布尔INP有合同关系,并由法国科学院赞助。
引用这篇文章: MAURIN Max (2024年3月9日), 抗生素,抗生素耐药性与环境, 环境百科全书,咨询于 2024年4月18日 [在线ISSN 2555-0950]网址: https://www.encyclopedie-environnement.org/zh/sante-zh/antibiotics-antibiotic-resistance-and-environment/.
环境百科全书中的文章是根据知识共享BY-NC-SA许可条款提供的,该许可授权复制的条件是:引用来源,不作商业使用,共享相同的初始条件,并且在每次重复使用或分发时复制知识共享BY-NC-SA许可声明。







