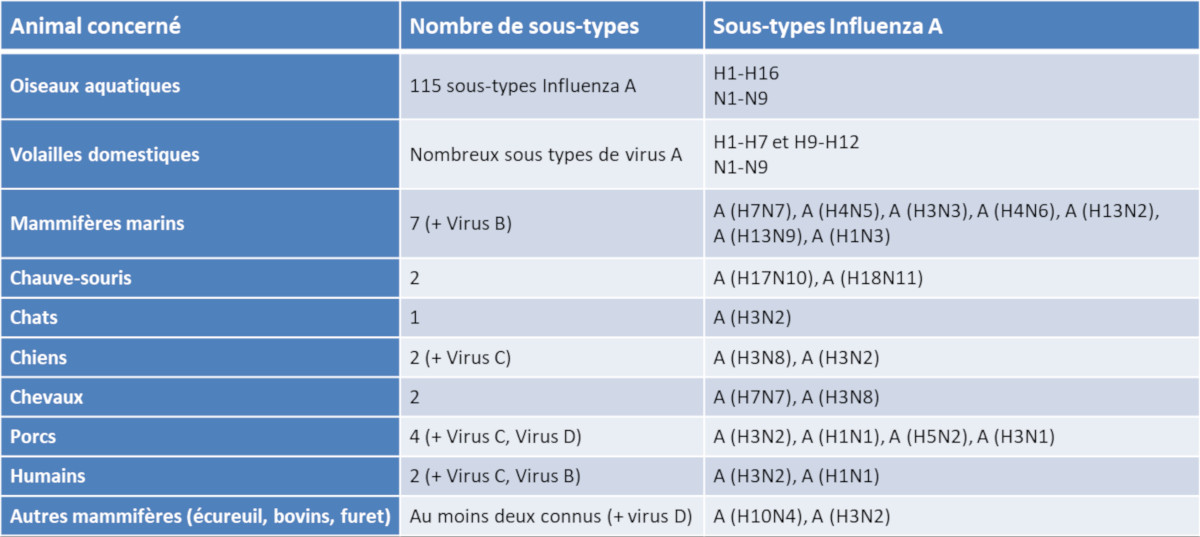
Les réassortiments génomiques : source majeure des pandémies grippales
PDFLes virus grippaux font partie de la famille des Orthomyxoviridae, caractérisé par une segmentation de leur génome ARN. Comme chez tous les virus à génome ARN, la mutation est une source importante de diversité de ces virus, due aux erreurs de réplication de leur enzyme (ARN polymérase). Les virus à génomes segmentés, comme le virus de la grippe, ont un autre mécanisme pour générer la diversité : le réassortiment.
- Les types A et B sont les principaux types qui se propagent régulièrement chez les humains et qui causent des épidémies de grippe saisonnière.
- La grippe B infecte presque exclusivement les humains et est moins fréquente que la grippe A. Les mutations se font environ de deux à trois fois plus lentement que celles de la grippe A. Étant donné que les humains sont l’hôte naturel de la grippe B, les virus de la grippe B ne créent pas de pandémie.
- Les virus de type C causent uniquement de légères infections respiratoires.
L’Organisation mondiale de la santé (OMS) a classé les virus grippaux de type A en sous-types en fonction des protéines de surface H et N, qui commandent respectivement l’entrée et la sortie des virus dans les cellules attaquées (cf tableau).
Tableau. Classification des virus grippaux de type A en sous-types en fonction des protéines de surface H et N selon l’Organisation mondiale de la santé (OMS).
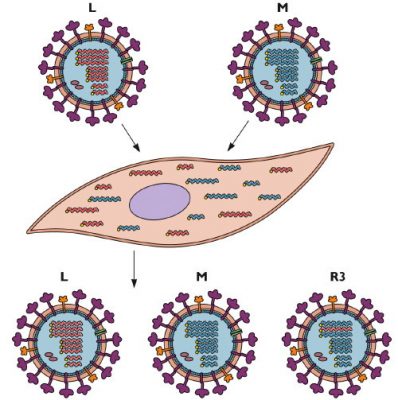
Si une cellule est infectée par deux souches virales différentes, les ARN des deux virus sont copiés dans le noyau. Lorsque de nouvelles particules virales sont assemblées au niveau de la membrane plasmique, chacun des 8 segments d’ARN peut provenir de l’un ou l’autre virus infectant. Les descendants qui héritent des ARN des deux parents sont appelés « réassortis ». C’est la situation que l’on peut observer au niveau pulmonaire chez le porc, dont les cellules possèdent les récepteurs à la fois de souches aviaires et de souches de mammifères. Ce processus est illustré dans le diagramme ci-dessous (Figure 2) [1], qui montre une cellule qui est co-infectée avec deux virus grippaux L et M. La cellule infectée produit à la fois des virus parentaux ainsi qu’un R3 réassorti qui hérite d’un segment d’ARN de la souche L et du reste de la souche M.
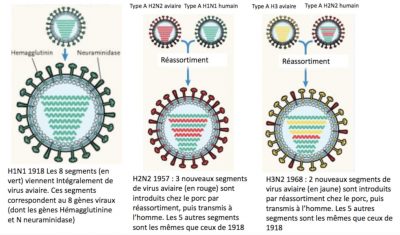
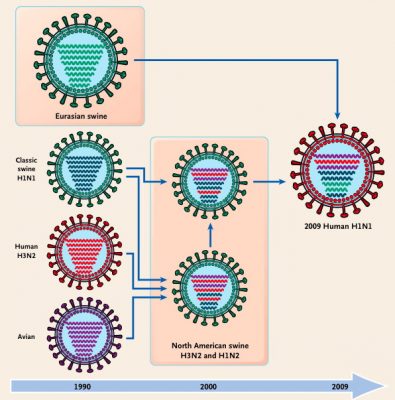
La souche pandémique H1N1 de 2009 est un réassorti des virus de la grippe aviaire, humaine et porcine, comme illustré en Figure 4 [1]. Le réassortiment ne peut se produire qu’entre virus de la grippe du même type. On ne comprend pas pourquoi les virus de la grippe A n’échangent jamais de segments d’ARN avec les virus de la grippe B ou C. Cependant, la raison est probablement liée au mécanisme d’assemblage viral (« packaging ») qui garantit que chaque virion grippal contient au moins une copie de chaque segment d’ARN.
En conclusion
Ces mécanismes de réassortiment, décrits sous le nom de sauts antigéniques, font émerger des souches radicalement nouvelles (au niveau antigénique), ce qui rend caduque l’immunité établie lors des épidémies précédentes. Enfin, pour qu’une pandémie à virus de grippe type A puisse se développer il est nécessaire qu’un tel virus réassorti acquière au minimum, par mutations, la capacité à reconnaître le récepteur humain pour se propager dans la population humaine.
Pour en savoir plus :
- Pandémies de grippe. Saison I: c’est quoi une pandémie ?
- Pandémies de grippe. Saison II: comment une pandémie de grippe peut survenir ?
- Trifonov, V., Khiabanian, H., & Rabadan, R. (2009). Geographic Dependence, Surveillance, and Origins of the 2009 Influenza A (H1N1) Virus New England Journal of Medicine DOI: 1056/NEJMp0904572
Références et notes
Image de couverture. Virus de la grippe en microscopie électronique. Crédit photo : Cynthia Goldsmith ; Content Providers(s): CDC/ Dr. Erskine. L. Palmer; Dr. M. L. Martin / Public domain
[1] Racaniello, V. The Virology Blog ; Reassortment of the influenza virus genome.
[2] Belshe, R.B. (2005) The Origins of Pandemic Influenza — Lessons from the 1918 Virus. N. Engl. J. Med. 353:2209-2211;
DOI: 10.1056/NEJMp058281